精彩导览
精彩导览
近日,物理科学与技术学院胡君辉教授/涂闪副教授光学研究团队,联合中国农业科学院热科所王俊刚副研究员、桂林电子科技大学张文涛教授和广西农业科学院甘蔗所刘昔辉研究员,共同完成的题为“High-precisionidentification transgenic sugarcane using active terahertz low-frequency excitation”的论文,在国际顶级学术期刊《Industrial Crops and Products》(中科院一区Top,2024年IF=5.60)在线发表。本研究提出了一种新颖、环保、快速且成本效益高的检测技术,结合了太赫兹(THz)时域光谱与等度量映射深度卷积神经网络(Isomap-DCNN)的机器学习模型,在识别五种转基因甘蔗品种样品时达到了100%的准确性。我校为论文第一完成单位,第一作者为涂闪副教授,通讯作者为王俊刚副研究员和刘昔辉研究员,第二、第三、第四、第五作者宋中宙、庞森灏、何起淋、苏靖凯为我院2022级在读本科生。

研究内容:甘蔗是热带和亚热带地区的主要经济作物,约占全球糖产量的75%。然而,生物应激源(如疾病、有害杂草和害虫)和非生物应激源(包括季节性季风失调、低温、盐度、碱度和土壤中的重金属污染),能显著影响甘蔗产量和品质。其中,施用除草剂被认为是田间控制作物产量和质量的有效方法。目前,草甘膦(GLY)和草铵膦(GLU)等除草剂属于非选择性广谱除草剂,具有成本低、毒性小、效率高等特点。本研究旨在解决传统化学检测方法在分类识别不同转基因甘蔗种类中所面临的污染大、成本高和耗时长等挑战,通过采集五种不同转基因甘蔗的THz吸光度光谱数据,并对原始光谱进行塔基滤波处理,分别应用t-SNE、LargeVis和Isomap降维方法,提取关键“指纹”特征。为提高模型的鲁棒性和泛化能力,采用了Kennard-Stone (KS)算法对训练集和测试集进行划分。最后将“指纹”特征矩阵输入到DT和DCNN分类器中进行分类。结果表明,Isomap-DCNN 模型在转基因甘蔗分类识别任务中表现出显著优势,其分类准确率达到100.00%。这表明,相较于t-SNE和LargeVis等注重局部结构的降维方法,保留全局结构的Isomap降维方法更适合提取具有流形结构的不同转基因甘蔗THz吸光度光谱数据的关键“指纹”特征。下面是部分结果与讨论复现:
|
|
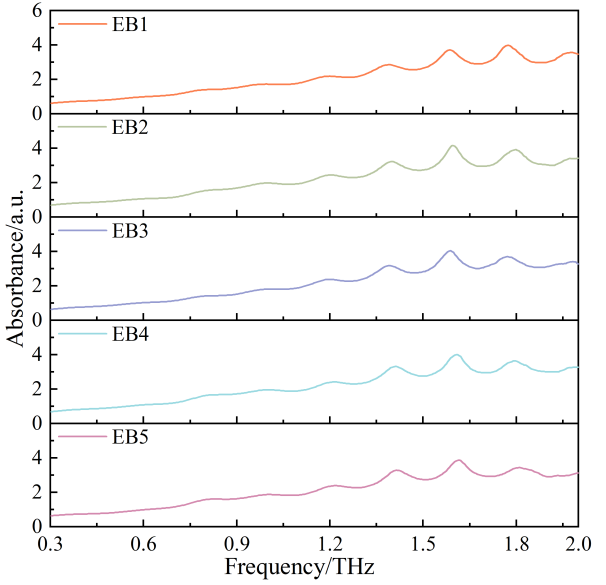
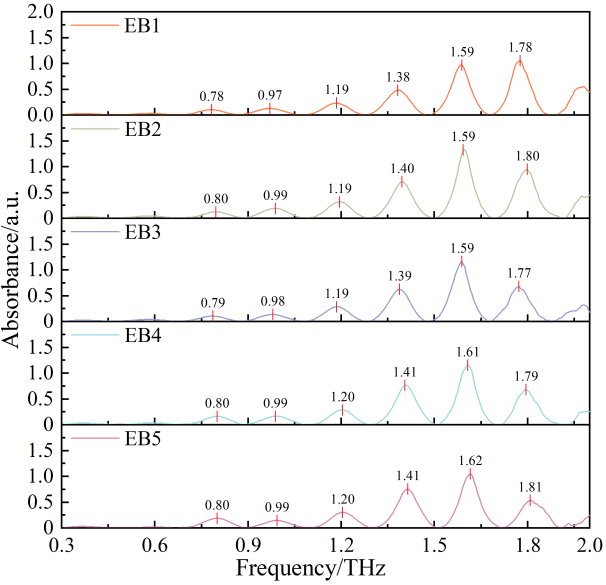
(a)五种转基因甘蔗THz吸光度光谱 | (b)基线校正处理的五种转基因甘蔗的THz吸光度光谱 |
图1五种转基因甘蔗的THz吸光度光谱可视化 | |
图1(a)给出了应用Tukey窗函数后五个转基因甘蔗品种的平均THz吸收光谱。图1(b)给出了对平均光谱进行基线校正增强THz吸收峰可视性后的图谱。五个转基因甘蔗品种的THz吸收光谱的总体模式是相似的,不同品种之间的峰形状和位置略有不同。其中,EB1的THz吸收光谱的峰位置明显不同于其他转基因甘蔗品种,而EB2、EB3、EB4和EB5在峰位置和值上仅表现出微小的差异。这对准确鉴定不同的转基因甘蔗提出了挑战。
|
|
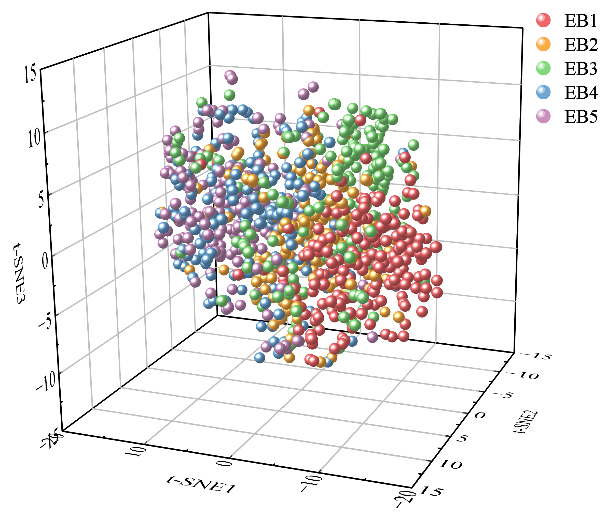
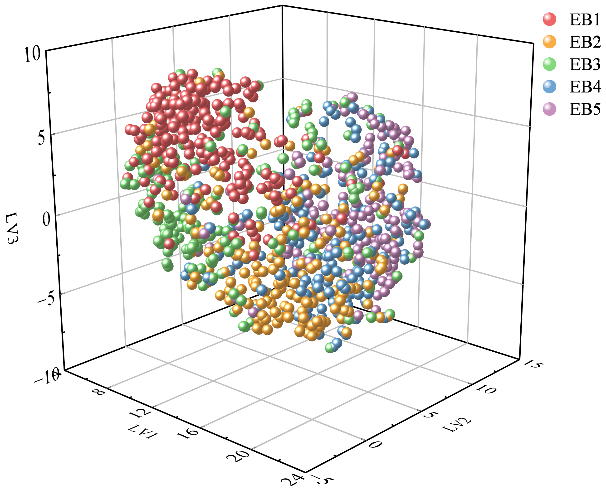
(a) t-SNE | (b) LV |
|
|
(c) Isomap | (d) 降维模型的轮廓系数 |
图2降维后的可视化结果 | |
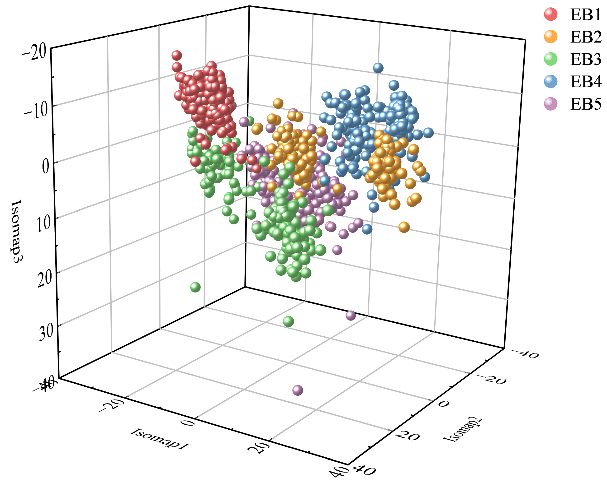
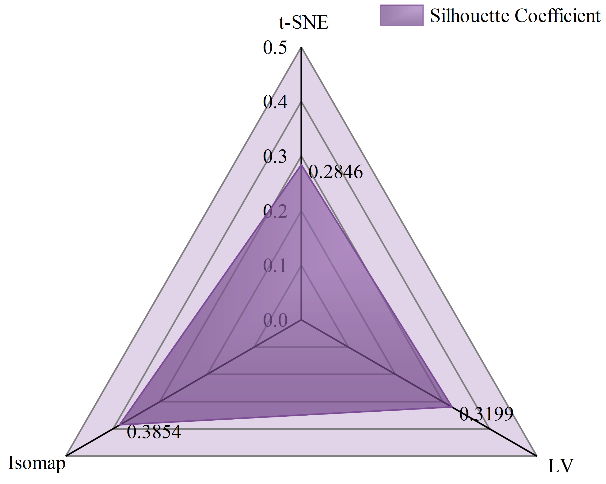
图2展示了不同转基因甘蔗经降维处理后的可视化结果。图2(a)展示了t-SNE降维的可视化,可以观察到类内样本点的聚集紧密度较低,且类间样品点的分离度不佳,边界模糊。这可能会影响对数据全局结构的准确表征,从而影响最终的分类效果。图2(b)展示了LV 降维的可视化结果,LV降维作为一种对t-SNE降维的优化,其整体降维效果与t-SNE相似,但在某些方面表现更佳。具体来说,LV降维在保持类内样本点的紧密度方面表现出与t-SNE相似的效果,但在类间样本点的分离效果上有所提升。特别是对于EB1样本点,LV降维初步实现了与其他四种转基因甘蔗样本点的分离,这一点相较于t-SNE降维是一个进步。图2(c)展示了Isomap降维的可视化,五种转基因甘蔗样品点初步实现了分离,不同种类样品点的类间距离明显增大,这增强了分类器的鲁棒性,能有效提高分类识别准确率。图2(d)显示了各降维方法的轮廓系数,其中,经t-SNE降维的数据的轮廓系数为0.285;经LV降维的数据的轮廓系数为0.320;经Isomap降维的数据的轮廓系数为0.385,为三种降维方法中最高。这表明,经Isomap降维的数据在类内紧密度和类间分离度方面效果最好,聚类质量最优。为了准确识别五种不同转基因甘蔗,有必要进一步采用机器学习和深度学习的模型对THz光谱数据集进行定性识别。
DT | DCNN | |
t-SNE |
|
|
LV |
|
|
Isomap |
|
|
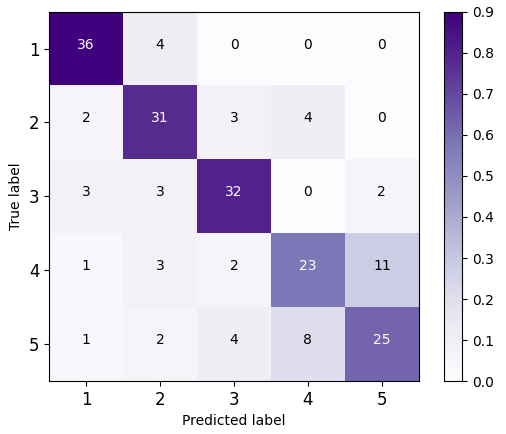
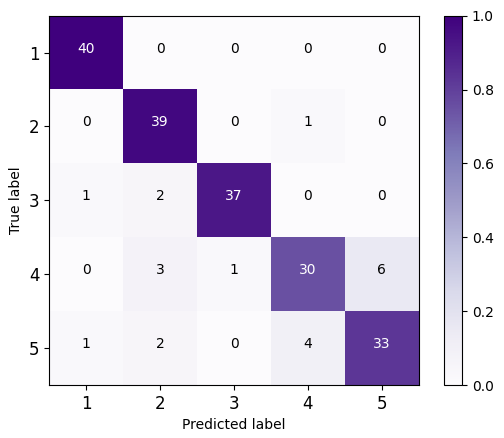
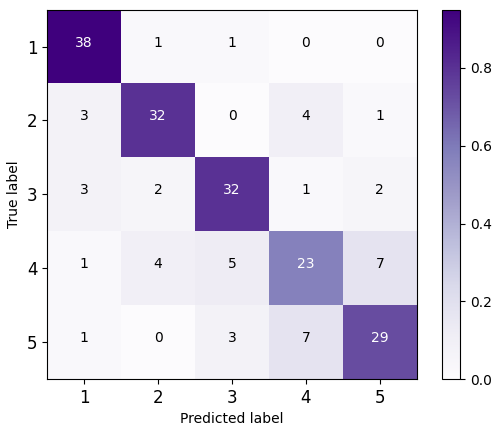
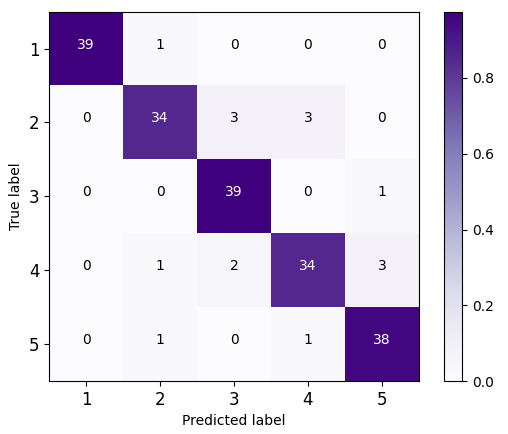
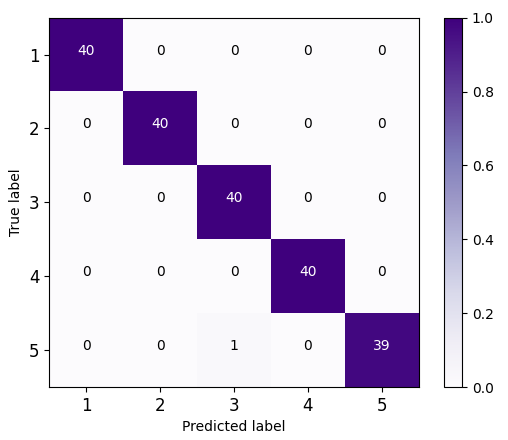
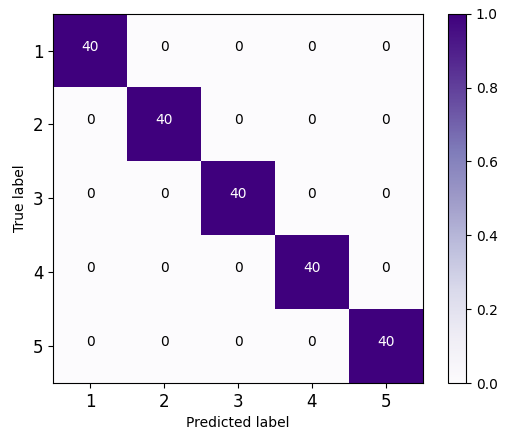
图3混淆矩阵 | ||
图3展示了六种模型的测试集分类混淆矩阵图。这一结果表明,通过采集转基因甘蔗的THz光谱数据,使用Isomap降维处理后,将THz“指纹”特征矩阵输入到DCNN分类器中,能高效、准确和无损的识别转基因甘蔗不同种类。
Intrinsic mode function | Sample set | Accuracy(%) |
t-SNE-DT | training | 78.00 |
prediction | 73.50 | |
t-SNE-DCNN | training | 96.38 |
prediction | 89.50 | |
LV-DT | training | 78.75 |
prediction | 77.00 | |
LV-DCNN | training | 94.63 |
prediction | 91.00 | |
Isomap-DT | training | 99.75 |
prediction | 99.50 | |
Isomap-DCNN | training | 100.00 |
prediction | 100.00 |
表1. 各模型的训练集与测试集分类准确率
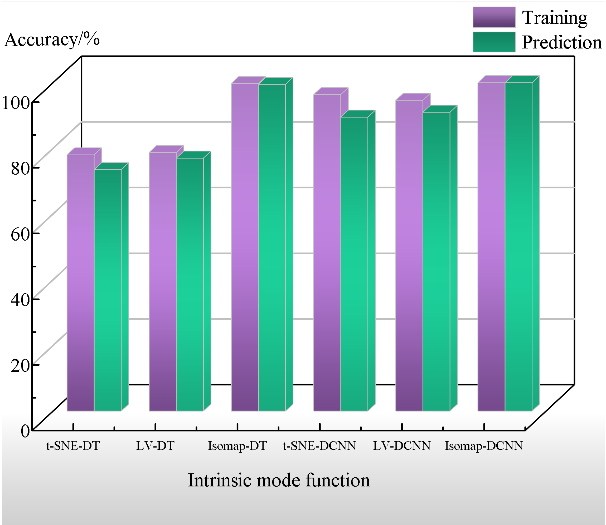
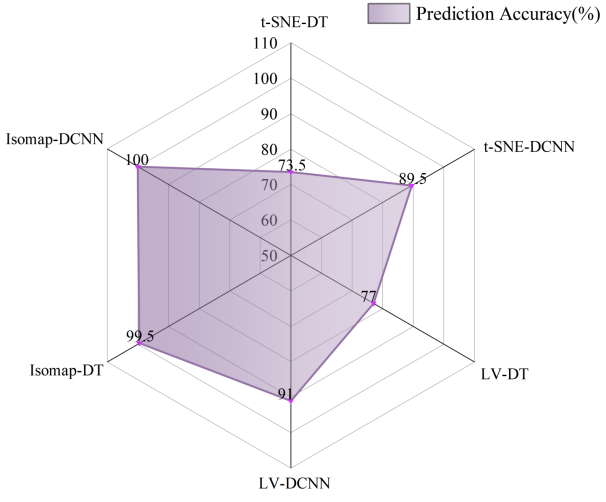
表1展示了6种分类模型训练集和测试集的分类准确率。六种模型的训练集准确率都高于测试集准确率,这代表着6种模型都在训练集上能够学习到数据的关键特征,有效捕捉到数据的结构,具有较好的泛化性能。其中,t-SNE-DT的分类准确率仅为73.50%,t-SNE-DCNN的分类准确率为89.50%,LV-DT的分类准确率仅为77.00%,LV-DCNN的分类准确率为91.00%,Isomap-DT的分类准确率达到了99.50%,Isomap-DCNN的分类准确率达到了完美的100.00%。
|
|
(a) 各模型训练集与测试集分类准确率柱形图 | (b) 各模型测试集分类准确率雷达图 |
图4. DT与DCNN分类器的性能对比 | |
图4直观展示了六种模型的整体性能对比。图4(a)为六种模型的训练集和测试集分类准确率的柱形图。柱形图显示,t-SNE与LV降维提取的THz“指纹”特征矩阵在DT和DCNN分类器中的训练集和测试集分类准确率差异较小,表明在转基因甘蔗分类任务中,t-SNE与LV的性能相似。而经过Isomap降维提取的THz“指纹”特征矩阵在DT和DCNN分类器中的分类准确率明显高于t-SNE和LV,这表明Isomap降维在转基因甘蔗分类任务中具有明显优势。图4(b)展示了六种模型的测试集分类准确率的雷达图。可以看出t-SNE-DCNN、LV-DCNN和Isomap-DNN模型的分类准确率均高于对应的t-SNE-DT、LV-DT和Isomap-DT模型的分类准确率。这表明基于深度学习的DCNN分类器在处理转基因甘蔗分类任务时,相较于传统机器学习DT分类器表现出更大的优势。
论文链接:https://doi.org/10.1016/j.indcrop.2025.120621